单核苷酸多态性 (SNPs)
介绍
每两个人之间大约每1000个碱基对就存在1个碱基对的差异。正是这些微小的基因变异造成了我们熟知的人类多样性——头发颜色、视力、药物过敏,甚至行为的差异。这些差异是由于单个核苷酸的变化引起的。
因此,单核苷酸多态性可以定义为由于单个核苷酸(腺嘌呤、胸腺嘧啶、胞嘧啶和鸟嘌呤)的改变而产生并在至少1%的人群中普遍存在的变异。这些SNPs作为分子标记,有助于诊断某些疾病。
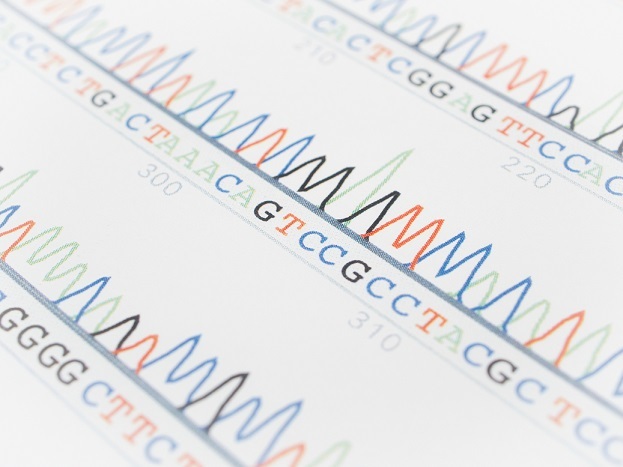
发生率
单核苷酸多态性可以存在于人的整个DNA中。平均而言,它们至少每1000个核苷酸就会出现一次。它们通常位于基因之间的DNA中,并充当分子标记。如果它们发生在基因或调控序列之间,则可能导致影响基因功能的某些疾病。
SNPs的类型
根据SNPs发现的区域及其影响,基本上有两种类型的SNPs:
非编码区SNPs。
编码区SNPs。
非编码区SNPs
之所以这样命名,是因为它们存在于非编码区或基因之间的区域。当SNPs存在于此类区域时,它可能不会影响蛋白质中氨基酸的序列,因为遗传密码是简并的。但它们在癌症期间可能会升高,并且在基因表达中也起着关键作用。
编码区SNPs
当它们存在于编码区时,它们可能改变也可能不改变氨基酸序列以及由此产生的蛋白质。根据这一点,编码区SNPs可以分为两种类型:
同义替换。
非同义替换。
同义替换
它们对氨基酸序列没有任何直接影响,因此不会影响最终的蛋白质,但它可能会显示其他一些变化。在细菌中,单个突变可能通过不合成任何蛋白质而只是导致膜折叠来帮助对抗许多药物的耐药性。
非同义替换
它们对蛋白质合成有直接影响。它们可能导致合成错误的蛋白质,或导致形成不成熟的蛋白质。根据此标准,有两种类型的非同义替换:
错义替换:编码区中单个核苷酸序列的任何变化都可能导致形成错误的蛋白质,其功能异常可能导致某些疾病或表型变化,例如早老症。
无义替换:核苷酸序列的变化可能由于在转录的mRNA上形成过早的终止密码子而导致形成过早的蛋白质,例如囊性纤维化。
SNPs分析
由于只有两个等位基因和三个基因型,SNPs可以通过多种方法进行分析,例如质谱、DNA测序、限制性片段长度多态性、毛细管电泳、杂交等。
SNPs的应用
通过聚类DNA序列或等位基因集,可以很容易地从单个SNP识别许多相互关联的SNPs。
SNP芯片和全基因组测序可用于生成全基因组遗传数据,这些数据反过来可用于识别与多种疾病、性状或表型相关的SNPs。所有这些都包含在全基因组关联研究中。由于这是基于全基因组研究,因此需要大样本量才能生成数据。
SNPs可用于研究与疾病或表型性状相关的基因变异。
SNPs用于研究从一个生物体传递到另一个生物体的基因簇,这种现象被称为遗传流行病学。
SNPs已广泛用于基于STR的DNA指纹识别,其中SNPs已被用于识别表型性状,如眼睛颜色、头发颜色等。但STRs比SNPs具有一定的优势,因为它们不依赖于数据库来进行样本的比较检查。
它们用于研究特定药物或药品的作用方式,也用于在治疗过程中测试该药物的有效性。这种在基因水平上研究药物作用的领域被称为药理基因组学。
编码区中的SNPs由于蛋白质发生改变而可能导致多种疾病。因此,对这些SNPs的详细研究可以深入了解多种疾病,例如2型糖尿病、自身免疫性疾病、精神疾病等。它们还有助于确定疾病治疗途径。
结论
大多数单核苷酸多态性对生物体的健康和发育没有任何重大影响。但在某些情况下,当它们是非同义的时,它们会产生遗传差异,这些差异可用于研究人类健康。
它们已被证明在与疾病发展风险、药物反应等相关的研究中非常重要。目前的研究领域包括植物育种,以开发对人类有用的新性状。


 数据结构
数据结构 网络
网络 关系数据库管理系统 (RDBMS)
关系数据库管理系统 (RDBMS) 操作系统
操作系统 Java
Java iOS
iOS HTML
HTML CSS
CSS Android
Android Python
Python C语言编程
C语言编程 C++
C++ C#
C# MongoDB
MongoDB MySQL
MySQL Javascript
Javascript PHP
PHP