
- Scikit-Learn 教程
- Scikit-Learn - 首页
- Scikit-Learn - 简介
- Scikit-Learn - 建模过程
- Scikit-Learn - 数据表示
- Scikit-Learn - 估计器 API
- Scikit-Learn - 约定
- Scikit-Learn - 线性建模
- Scikit-Learn - 扩展线性建模
- 随机梯度下降
- Scikit-Learn - 支持向量机
- Scikit-Learn - 异常检测
- Scikit-Learn - K 近邻算法
- Scikit-Learn - KNN 学习
- 朴素贝叶斯分类
- Scikit-Learn - 决策树
- 随机决策树
- Scikit-Learn - 集成方法
- Scikit-Learn - 聚类方法
- 聚类性能评估
- 使用 PCA 进行降维
- Scikit-Learn 有用资源
- Scikit-Learn - 快速指南
- Scikit-Learn - 有用资源
- Scikit-Learn - 讨论
Scikit-Learn - 支持向量机
本章讨论一种称为支持向量机 (SVM) 的机器学习方法。
介绍
支持向量机 (SVM) 是一种强大而灵活的监督式机器学习方法,用于分类、回归和异常值检测。SVM 在高维空间中非常有效,通常用于分类问题。SVM 广受欢迎且内存效率高,因为它们在决策函数中使用了训练点的子集。
SVM 的主要目标是将数据集划分为多个类别,以便找到一个**最大间隔超平面 (MMH)**,这可以通过以下两个步骤完成:
支持向量机将首先迭代生成超平面,以最佳方式分离类别。
之后,它将选择正确分离类别的超平面。
SVM 中的一些重要概念如下:
**支持向量** - 可以定义为最靠近超平面的数据点。支持向量有助于确定分离线。
**超平面** - 分割具有不同类别的对象集的决策平面或空间。
**间隔** - 不同类别最靠近数据点之间的两条线之间的间隙称为间隔。
以下图表将让您深入了解这些 SVM 概念:
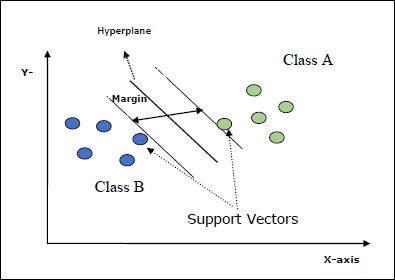
Scikit-learn 中的 SVM 支持稀疏和密集样本向量作为输入。
SVM 的分类
Scikit-learn 提供了三个类,即**SVC、NuSVC** 和**LinearSVC**,它们可以执行多类分类。
SVC
它是 C 支持向量分类,其实现基于**libsvm**。Scikit-learn 使用的模块是**sklearn.svm.SVC**。此类根据一对一方案处理多类支持。
参数
下表包含**sklearn.svm.SVC** 类使用的参数:
| 序号 | 参数及描述 |
|---|---|
| 1 |
C - float,可选,默认值 = 1.0 它是误差项的惩罚参数。 |
| 2 |
kernel - string,可选,默认值 = 'rbf' 此参数指定算法中要使用的内核类型。我们可以从以下选项中选择一个:**'linear'、'poly'、'rbf'、'sigmoid'、'precomputed'**。内核的默认值为**'rbf'**。 |
| 3 |
degree - int,可选,默认值 = 3 它表示“poly”内核函数的度数,其他所有内核都会忽略它。 |
| 4 |
gamma - {'scale'、'auto'} 或 float, 它是内核“rbf”、“poly”和“sigmoid”的内核系数。 |
| 5 |
可选默认值 - = 'scale' 如果您选择默认值,即 gamma = 'scale',则 SVC 使用的 gamma 值为 1/(𝑛_𝑓𝑒𝑎𝑡𝑢𝑟𝑒𝑠∗𝑋.𝑣𝑎𝑟())。 另一方面,如果 gamma = 'auto',则它使用 1/𝑛_𝑓𝑒𝑎𝑡𝑢𝑟𝑒𝑠。 |
| 6 |
coef0 - float,可选,默认值 = 0.0 内核函数中的一个独立项,仅在“poly”和“sigmoid”中有效。 |
| 7 |
tol - float,可选,默认值 = 1.e-3 此参数表示迭代的停止条件。 |
| 8 |
shrinking - Boolean,可选,默认值 = True 此参数表示我们是否要使用收缩启发式方法。 |
| 9 |
verbose - Boolean,默认值:false 它启用或禁用详细输出。其默认值为 false。 |
| 10 |
probability - boolean,可选,默认值 = true 此参数启用或禁用概率估计。默认值为 false,但在调用 fit 之前必须启用它。 |
| 11 |
max_iter - int,可选,默认值 = -1 顾名思义,它表示求解器中的最大迭代次数。值 -1 表示迭代次数没有限制。 |
| 12 |
cache_size - float,可选 此参数将指定内核缓存的大小。该值将以 MB(兆字节)为单位。 |
| 13 |
random_state - int、RandomState 实例或 None,可选,默认值 = none 此参数表示用于在洗牌数据时生成的伪随机数的种子。选项如下:
|
| 14 |
class_weight - {dict、'balanced'},可选 此参数将类 j 的参数 C 设置为 𝑐𝑙𝑎𝑠𝑠_𝑤𝑒𝑖𝑔ℎ𝑡[𝑗]∗𝐶 用于 SVC。如果我们使用默认选项,则意味着所有类都应该具有权重 1。另一方面,如果您选择class_weight:balanced,它将使用 y 的值来自动调整权重。 |
| 15 |
decision_function_shape - 'ovo'、'ovr',默认值 = 'ovr' 此参数将决定算法是否返回形状为所有其他分类器的'ovr'(一对多)决策函数,或 libsvm 的原始ovo(一对一)决策函数。 |
| 16 |
break_ties - boolean,可选,默认值 = false True - predict 将根据 decision_function 的置信度值打破平局 False - predict 将返回平局类中的第一个类。 |
属性
下表包含**sklearn.svm.SVC** 类使用的属性:
| 序号 | 属性及描述 |
|---|---|
| 1 |
support_ - 类似数组,形状 = [n_SV] 它返回支持向量的索引。 |
| 2 |
support_vectors_ - 类似数组,形状 = [n_SV, n_features] 它返回支持向量。 |
| 3 |
n_support_ - 类似数组,dtype=int32,形状 = [n_class] 它表示每个类的支持向量数量。 |
| 4 |
dual_coef_ - 数组,形状 = [n_class-1,n_SV] 这些是决策函数中支持向量的系数。 |
| 5 |
coef_ - 数组,形状 = [n_class * (n_class-1)/2, n_features] 此属性仅在使用线性内核时可用,它提供了分配给特征的权重。 |
| 6 |
intercept_ - 数组,形状 = [n_class * (n_class-1)/2] 它表示决策函数中的独立项(常数)。 |
| 7 |
fit_status_ - int 如果正确拟合,则输出为 0。如果拟合不正确,则输出为 1。 |
| 8 |
classes_ - 形状为 [n_classes] 的数组 它给出类的标签。 |
实现示例
与其他分类器一样,SVC 也必须使用以下两个数组进行拟合:
包含训练样本的数组X。其大小为 [n_samples, n_features]。
包含训练样本的目标值,即类标签的数组Y。其大小为 [n_samples]。
以下 Python 脚本使用sklearn.svm.SVC 类:
import numpy as np X = np.array([[-1, -1], [-2, -1], [1, 1], [2, 1]]) y = np.array([1, 1, 2, 2]) from sklearn.svm import SVC SVCClf = SVC(kernel = 'linear',gamma = 'scale', shrinking = False,) SVCClf.fit(X, y)
输出
SVC(C = 1.0, cache_size = 200, class_weight = None, coef0 = 0.0, decision_function_shape = 'ovr', degree = 3, gamma = 'scale', kernel = 'linear', max_iter = -1, probability = False, random_state = None, shrinking = False, tol = 0.001, verbose = False)
示例
现在,一旦拟合,我们就可以借助以下 Python 脚本获取权重向量:
SVCClf.coef_
输出
array([[0.5, 0.5]])
示例
类似地,我们可以获取其他属性的值,如下所示:
SVCClf.predict([[-0.5,-0.8]])
输出
array([1])
示例
SVCClf.n_support_
输出
array([1, 1])
示例
SVCClf.support_vectors_
输出
array(
[
[-1., -1.],
[ 1., 1.]
]
)
示例
SVCClf.support_
输出
array([0, 2])
示例
SVCClf.intercept_
输出
array([-0.])
示例
SVCClf.fit_status_
输出
0
NuSVC
NuSVC 是 Nu 支持向量分类。它是 Scikit-learn 提供的另一个可以执行多类分类的类。它类似于 SVC,但 NuSVC 接受略微不同的参数集。与 SVC 不同的参数如下:
nu - float,可选,默认值 = 0.5
它表示训练误差分数的上限和支持向量分数的下限。其值应在 (o,1] 的区间内。
其余参数和属性与 SVC 相同。
实现示例
我们也可以使用sklearn.svm.NuSVC 类实现相同的示例。
import numpy as np X = np.array([[-1, -1], [-2, -1], [1, 1], [2, 1]]) y = np.array([1, 1, 2, 2]) from sklearn.svm import NuSVC NuSVCClf = NuSVC(kernel = 'linear',gamma = 'scale', shrinking = False,) NuSVCClf.fit(X, y)
输出
NuSVC(cache_size = 200, class_weight = None, coef0 = 0.0, decision_function_shape = 'ovr', degree = 3, gamma = 'scale', kernel = 'linear', max_iter = -1, nu = 0.5, probability = False, random_state = None, shrinking = False, tol = 0.001, verbose = False)
我们可以像在 SVC 中一样获取其余属性的输出。
LinearSVC
它是线性支持向量分类。它类似于具有 kernel = 'linear' 的 SVC。它们之间的区别在于LinearSVC 是根据 liblinear 实现的,而 SVC 是在libsvm 中实现的。这就是LinearSVC 在惩罚和损失函数的选择方面具有更大灵活性的原因。它还可以更好地扩展到大量样本。
如果我们谈论它的参数和属性,那么它不支持'kernel',因为它被假定为线性,并且它也缺少一些属性,例如support_、support_vectors_、n_support_、fit_status_ 和dual_coef_。
但是,它支持penalty 和loss 参数,如下所示:
penalty - string,L1 或 L2(默认值 = 'L2')
此参数用于指定惩罚(正则化)中使用的范数(L1 或 L2)。
loss - string,hinge、squared_hinge(默认值 = squared_hinge)
它表示损失函数,其中“hinge”是标准 SVM 损失,“squared_hinge”是 hinge 损失的平方。
实现示例
以下 Python 脚本使用sklearn.svm.LinearSVC 类:
from sklearn.svm import LinearSVC from sklearn.datasets import make_classification X, y = make_classification(n_features = 4, random_state = 0) LSVCClf = LinearSVC(dual = False, random_state = 0, penalty = 'l1',tol = 1e-5) LSVCClf.fit(X, y)
输出
LinearSVC(C = 1.0, class_weight = None, dual = False, fit_intercept = True, intercept_scaling = 1, loss = 'squared_hinge', max_iter = 1000, multi_class = 'ovr', penalty = 'l1', random_state = 0, tol = 1e-05, verbose = 0)
示例
现在,一旦拟合,模型就可以预测新值,如下所示:
LSVCClf.predict([[0,0,0,0]])
输出
[1]
示例
对于上述示例,我们可以借助以下 Python 脚本获取权重向量:
LSVCClf.coef_
输出
[[0. 0. 0.91214955 0.22630686]]
示例
类似地,我们可以借助以下 Python 脚本获取截距的值:
LSVCClf.intercept_
输出
[0.26860518]
SVM 回归
如前所述,SVM 用于分类和回归问题。Scikit-learn 的支持向量分类 (SVC) 方法也可以扩展到解决回归问题。该扩展方法称为支持向量回归 (SVR)。
SVM 和 SVR 之间的基本相似性
SVC 创建的模型仅依赖于训练数据的一个子集。为什么?因为构建模型的成本函数不关心位于间隔之外的训练数据点。
而 SVR(支持向量回归)生成的模型也仅依赖于训练数据的一个子集。为什么?因为构建模型的成本函数会忽略任何靠近模型预测的训练数据点。
Scikit-learn 提供了三个类,即**SVR、NuSVR 和 LinearSVR**,作为 SVR 的三种不同实现。
SVR
它是 Epsilon 支持向量回归,其实现基于**libsvm**。与SVC 相反,模型中有两个自由参数,即'C' 和'epsilon'。
epsilon - float,可选,默认值 = 0.1
它表示 epsilon-SVR 模型中的 epsilon,并指定 epsilon 管,在该管内,在训练损失函数中,对预测值与实际值之间的距离在 epsilon 内的点不关联任何惩罚。
其余参数和属性与我们在SVC 中使用的类似。
实现示例
以下 Python 脚本使用sklearn.svm.SVR 类:
from sklearn import svm X = [[1, 1], [2, 2]] y = [1, 2] SVRReg = svm.SVR(kernel = ’linear’, gamma = ’auto’) SVRReg.fit(X, y)
输出
SVR(C = 1.0, cache_size = 200, coef0 = 0.0, degree = 3, epsilon = 0.1, gamma = 'auto', kernel = 'linear', max_iter = -1, shrinking = True, tol = 0.001, verbose = False)
示例
现在,一旦拟合,我们就可以借助以下 Python 脚本获取权重向量:
SVRReg.coef_
输出
array([[0.4, 0.4]])
示例
类似地,我们可以获取其他属性的值,如下所示:
SVRReg.predict([[1,1]])
输出
array([1.1])
类似地,我们也可以获取其他属性的值。
NuSVR
NuSVR 是 Nu 支持向量回归。它类似于 NuSVC,但 NuSVR 使用参数nu来控制支持向量的数量。此外,与 NuSVC 中nu替换 C 参数不同,这里它替换了epsilon。
实现示例
以下 Python 脚本使用sklearn.svm.SVR 类:
from sklearn.svm import NuSVR import numpy as np n_samples, n_features = 20, 15 np.random.seed(0) y = np.random.randn(n_samples) X = np.random.randn(n_samples, n_features) NuSVRReg = NuSVR(kernel = 'linear', gamma = 'auto',C = 1.0, nu = 0.1)^M NuSVRReg.fit(X, y)
输出
NuSVR(C = 1.0, cache_size = 200, coef0 = 0.0, degree = 3, gamma = 'auto', kernel = 'linear', max_iter = -1, nu = 0.1, shrinking = True, tol = 0.001, verbose = False)
示例
现在,一旦拟合,我们就可以借助以下 Python 脚本获取权重向量:
NuSVRReg.coef_
输出
array(
[
[-0.14904483, 0.04596145, 0.22605216, -0.08125403, 0.06564533,
0.01104285, 0.04068767, 0.2918337 , -0.13473211, 0.36006765,
-0.2185713 , -0.31836476, -0.03048429, 0.16102126, -0.29317051]
]
)
类似地,我们也可以获取其他属性的值。
LinearSVR
它是线性支持向量回归。它类似于具有 kernel = 'linear' 的 SVR。它们之间的区别在于LinearSVR是用liblinear实现的,而 SVC是用libsvm实现的。这就是LinearSVR在惩罚和损失函数的选择方面具有更多灵活性的原因。它也更适合于大量样本。
如果我们谈论它的参数和属性,那么它不支持'kernel',因为它被假定为线性,并且它也缺少一些属性,例如support_、support_vectors_、n_support_、fit_status_ 和dual_coef_。
但是,它支持以下'loss'参数:
loss - 字符串,可选,默认值 = 'epsilon_insensitive'
它表示损失函数,其中 epsilon_insensitive 损失是 L1 损失,而平方 epsilon_insensitive 损失是 L2 损失。
实现示例
以下 Python 脚本使用sklearn.svm.LinearSVR类:
from sklearn.svm import LinearSVR from sklearn.datasets import make_regression X, y = make_regression(n_features = 4, random_state = 0) LSVRReg = LinearSVR(dual = False, random_state = 0, loss = 'squared_epsilon_insensitive',tol = 1e-5) LSVRReg.fit(X, y)
输出
LinearSVR( C=1.0, dual=False, epsilon=0.0, fit_intercept=True, intercept_scaling=1.0, loss='squared_epsilon_insensitive', max_iter=1000, random_state=0, tol=1e-05, verbose=0 )
示例
现在,一旦拟合,模型就可以预测新值,如下所示:
LSRReg.predict([[0,0,0,0]])
输出
array([-0.01041416])
示例
对于上述示例,我们可以借助以下 Python 脚本获取权重向量:
LSRReg.coef_
输出
array([20.47354746, 34.08619401, 67.23189022, 87.47017787])
示例
类似地,我们可以借助以下 Python 脚本获取截距的值:
LSRReg.intercept_
输出
array([-0.01041416])