
- 大数据分析教程
- 大数据分析 - 首页
- 大数据分析 - 概述
- 大数据分析 - 特性
- 大数据分析 - 数据生命周期
- 大数据分析 - 架构
- 大数据分析 - 方法论
- 大数据分析 - 核心交付成果
- 大数据采用与规划考虑
- 大数据分析 - 主要利益相关者
- 大数据分析 - 数据分析师
- 大数据分析 - 数据科学家
- 大数据分析有用资源
- 大数据分析 - 快速指南
- 大数据分析 - 资源
- 大数据分析 - 讨论
大数据分析 - 统计方法
在分析数据时,可以使用统计方法。执行基本分析所需的常用工具包括:
- 相关性分析
- 方差分析
- 假设检验
处理大型数据集时,这些方法不会造成问题,因为除了相关性分析外,这些方法在计算上并不密集。在这种情况下,始终可以抽取样本,结果应该稳健。
相关性分析
相关性分析旨在寻找数值变量之间的线性关系。这在不同情况下都很有用。一种常见用途是探索性数据分析,书中16.0.2节有一个这种方法的基本示例。首先,上述示例中使用的相关性指标基于**皮尔逊系数**。然而,还有一个有趣的相关性指标不受异常值的影响。这个指标称为斯皮尔曼相关性。
**斯皮尔曼相关性**指标比皮尔逊方法更能抵抗异常值的影响,并且在数据不服从正态分布时,能更好地估计数值变量之间的线性关系。
library(ggplot2)
# Select variables that are interesting to compare pearson and spearman
correlation methods.
x = diamonds[, c('x', 'y', 'z', 'price')]
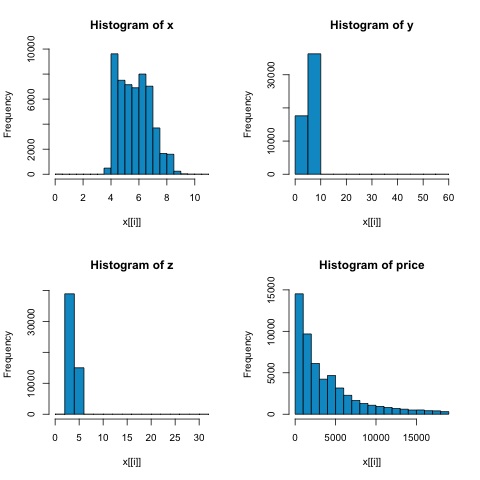
# From the histograms we can expect differences in the correlations of both
metrics.
# In this case as the variables are clearly not normally distributed, the
spearman correlation
# is a better estimate of the linear relation among numeric variables.
par(mfrow = c(2,2))
colnm = names(x)
for(i in 1:4) {
hist(x[[i]], col = 'deepskyblue3', main = sprintf('Histogram of %s', colnm[i]))
}
par(mfrow = c(1,1))
从下图中的直方图可以看出,两种指标的相关性存在差异。在这种情况下,由于变量显然不服从正态分布,因此斯皮尔曼相关性是数值变量之间线性关系的更好估计。
为了在R中计算相关性,请打开包含此代码部分的文件**bda/part2/statistical_methods/correlation/correlation.R**。
## Correlation Matrix - Pearson and spearman cor_pearson <- cor(x, method = 'pearson') cor_spearman <- cor(x, method = 'spearman') ### Pearson Correlation print(cor_pearson) # x y z price # x 1.0000000 0.9747015 0.9707718 0.8844352 # y 0.9747015 1.0000000 0.9520057 0.8654209 # z 0.9707718 0.9520057 1.0000000 0.8612494 # price 0.8844352 0.8654209 0.8612494 1.0000000 ### Spearman Correlation print(cor_spearman) # x y z price # x 1.0000000 0.9978949 0.9873553 0.9631961 # y 0.9978949 1.0000000 0.9870675 0.9627188 # z 0.9873553 0.9870675 1.0000000 0.9572323 # price 0.9631961 0.9627188 0.9572323 1.0000000
卡方检验
卡方检验允许我们检验两个随机变量是否独立。这意味着每个变量的概率分布都不会影响另一个变量。为了在R中评估检验,我们首先需要创建一个列联表,然后将该表传递给**chisq.test R**函数。
例如,让我们检查diamonds数据集中的cut和color变量之间是否存在关联。该检验正式定义为:
- H0:变量cut和diamond是独立的
- H1:变量cut和diamond不是独立的
从变量名称来看,我们假设这两个变量之间存在关系,但检验可以提供一个客观的“规则”,说明此结果是否显著。
在下面的代码片段中,我们发现检验的p值为2.2e-16,实际上几乎为零。然后在进行**蒙特卡罗模拟**后运行检验,我们发现p值为0.0004998,仍然远低于阈值0.05。此结果意味着我们拒绝零假设(H0),因此我们认为变量**cut**和**color**不是独立的。
library(ggplot2) # Use the table function to compute the contingency table tbl = table(diamonds$cut, diamonds$color) tbl # D E F G H I J # Fair 163 224 312 314 303 175 119 # Good 662 933 909 871 702 522 307 # Very Good 1513 2400 2164 2299 1824 1204 678 # Premium 1603 2337 2331 2924 2360 1428 808 # Ideal 2834 3903 3826 4884 3115 2093 896 # In order to run the test we just use the chisq.test function. chisq.test(tbl) # Pearson’s Chi-squared test # data: tbl # X-squared = 310.32, df = 24, p-value < 2.2e-16 # It is also possible to compute the p-values using a monte-carlo simulation # It's needed to add the simulate.p.value = TRUE flag and the amount of simulations chisq.test(tbl, simulate.p.value = TRUE, B = 2000) # Pearson’s Chi-squared test with simulated p-value (based on 2000 replicates) # data: tbl # X-squared = 310.32, df = NA, p-value = 0.0004998
t检验
**t检验**的目的是评估名义变量的不同组之间数值变量的分布是否存在差异。为了演示这一点,我将选择因子变量cut的Fair和Ideal水平,然后我们将比较这两个组之间数值变量的值。
data = diamonds[diamonds$cut %in% c('Fair', 'Ideal'), ]
data$cut = droplevels.factor(data$cut) # Drop levels that aren’t used from the
cut variable
df1 = data[, c('cut', 'price')]
# We can see the price means are different for each group
tapply(df1$price, df1$cut, mean)
# Fair Ideal
# 4358.758 3457.542
t检验在R中使用**t.test**函数实现。t.test的公式接口是最简单的使用方法,其思想是用组变量解释数值变量。
例如:**t.test(numeric_variable ~ group_variable, data = data)**。在前面的示例中,**numeric_variable**是**price**,**group_variable**是**cut**。
从统计学的角度来看,我们正在检验数值变量在两组之间的分布是否存在差异。正式的假设检验用零假设(H0)和备择假设(H1)来描述。
H0:Fair和Ideal组之间price变量的分布没有差异
H1:Fair和Ideal组之间price变量的分布存在差异
以下可以在R中使用以下代码实现:
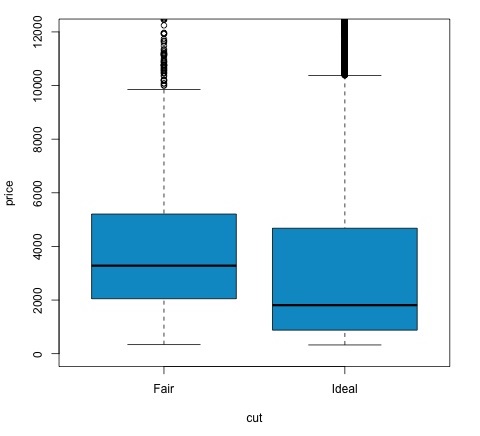
t.test(price ~ cut, data = data) # Welch Two Sample t-test # # data: price by cut # t = 9.7484, df = 1894.8, p-value < 2.2e-16 # alternative hypothesis: true difference in means is not equal to 0 # 95 percent confidence interval: # 719.9065 1082.5251 # sample estimates: # mean in group Fair mean in group Ideal # 4358.758 3457.542 # Another way to validate the previous results is to just plot the distributions using a box-plot plot(price ~ cut, data = data, ylim = c(0,12000), col = 'deepskyblue3')
我们可以通过检查p值是否小于0.05来分析检验结果。如果是这种情况,我们保留备择假设。这意味着我们在cut因子的两个水平之间发现了价格差异。从水平的名称来看,我们本可以预期这个结果,但我们不会预期Fail组的平均价格会高于Ideal组。我们可以通过比较每个因子的均值来观察这一点。
**plot**命令生成的图形显示了价格和cut变量之间的关系。这是一个箱线图;我们在16.0.1节中介绍过这种图,但它基本上显示了我们正在分析的cut的两个水平的price变量的分布。
方差分析
方差分析(ANOVA)是一种统计模型,用于通过比较每组的均值和方差来分析组分布之间的差异,该模型由罗纳德·费舍尔开发。ANOVA提供了一个统计检验,用于检验多个组的均值是否相等,因此它将t检验推广到多于两组的情况。
ANOVA对于比较三个或更多组的统计显着性很有用,因为进行多次双样本t检验会导致犯I型统计错误的可能性增加。
在提供数学解释方面,需要了解以下内容才能理解该检验。
xij = x + (xi − x) + (xij − x)
这导致以下模型:
xij = μ + αi + ∈ij
其中μ是总均值,αi是第i个组均值。误差项∈ij假设来自正态分布的iid。检验的零假设是:
α1 = α2 = … = αk
在计算检验统计量方面,我们需要计算两个值:
- 组间差异的平方和:
$$SSD_B = \sum_{i}^{k} \sum_{j}^{n}(\bar{x_{\bar{i}}} - \bar{x})^2$$
- 组内平方和
$$SSD_W = \sum_{i}^{k} \sum_{j}^{n}(\bar{x_{\bar{ij}}} - \bar{x_{\bar{i}}})^2$$
其中SSDB的自由度为k−1,SSDW的自由度为N−k。然后我们可以定义每个指标的均方差。
MSB = SSDB / (k - 1)
MSw = SSDw / (N - k)
最后,ANOVA中的检验统计量定义为上述两个量的比率
F = MSB / MSw
它服从自由度为k−1和N−k的F分布。如果零假设为真,F值可能接近1。否则,组间均方MSB可能很大,这将导致较大的F值。
基本上,ANOVA检查总方差的两个来源,并查看哪个部分的贡献更大。这就是为什么它被称为方差分析,尽管目的是比较组均值。
在计算统计量方面,在R中实际上相当简单。以下示例将演示如何操作以及如何绘制结果。
library(ggplot2)
# We will be using the mtcars dataset
head(mtcars)
# mpg cyl disp hp drat wt qsec vs am gear carb
# Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
# Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
# Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
# Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
# Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
# Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
# Let's see if there are differences between the groups of cyl in the mpg variable.
data = mtcars[, c('mpg', 'cyl')]
fit = lm(mpg ~ cyl, data = mtcars)
anova(fit)
# Analysis of Variance Table
# Response: mpg
# Df Sum Sq Mean Sq F value Pr(>F)
# cyl 1 817.71 817.71 79.561 6.113e-10 ***
# Residuals 30 308.33 10.28
# Signif. codes: 0 *** 0.001 ** 0.01 * 0.05 .
# Plot the distribution
plot(mpg ~ as.factor(cyl), data = mtcars, col = 'deepskyblue3')
代码将产生以下输出:
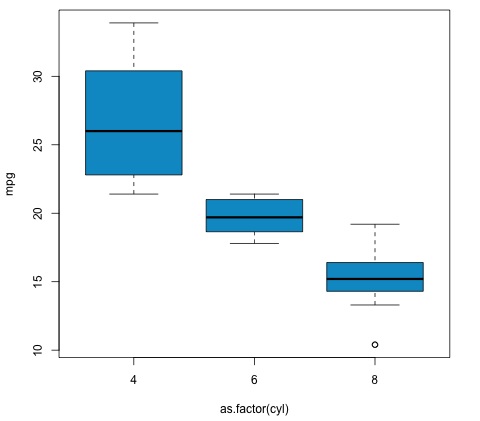
我们在示例中获得的p值远小于0.05,因此R返回符号'***'来表示这一点。这意味着我们拒绝零假设,并且我们在cyl变量的不同组之间发现了mpg均值的差异。